Las cepas probióticas nativas son elegidas para la producción de productos lácteos funcionales debido a que presentan ventajas tecnológicas. La selección preliminar se basa en ensayos in vitro que predicen su seguridad y propiedades funcionales. El objetivo del presente estudio fue evaluar in vitro la sensibilidad a antibióticos, el pasaje por el tracto gastrointestinal y la actividad antimicrobiana de la cepa nativa Lactococcus lactis GU967439, aislada de leche cruda proveniente de una quesería artesanal de Colonia, Uruguay. Se utilizó a L. lactis ATCC N° 11454 como referencia. L. lactis GU967439 fue sensible a todos los antibióticos activos frente a Gram positivos. Además, fue resistente a la simulación del jugo gástrico a pH 3, pancreatina y bilis. El sobrenadante libre de células de L. lactis GU967439 inhibió el crecimiento de Staphylococcus aureus ATCC 6538, Staphylococcus aureus ATCC 29213, Enterococcus faecalis ATCC 29212, Listeria innocua ATCC 33090, Streptococcus bovis 2.5 (WT) y presentó mejor actividad que la cepa L. lactis ATCC N° 11454. Sin embargo, ninguna de las cepas inhibió a los microorganismos Gram negativos evaluados (Escherichia coli ATCC 25922, Pseudomonas aeruginosa ATCC 9027). Estos resultados dan base a futuros trabajos que permitan continuar caracterizando el potencial probiótico de L. lactis GU967439.
Palabras clave: bacterias ácidolácticas, resistencia gastrointestinal, actividad antimicrobiana, bacteriocinas, resistencia a antibióticos.
As cepas probióticas nativas são escolhidas para a produção de derivados lácteos funcionais devido às vantagens tecnológicas que apresentam. A seleção preliminar é baseada em ensaios in vitro que predizem a sua segurança e também as propriedades funcionais. O objetivo do presente estudo foi avaliar in vitro a sensibilidade aos antibióticos, a passagem pelo trato gastrointestinal e a atividade antimicrobiana da cepa nativa Lactococcus lactis GU967439, isolada do leite cru proveniente de um estabelecimento artesanal em Colonia, Uruguai. L. lactis ATCC N° 11454 foi utilizada como referência. A cepa L. lactis GU967439 foi sensível a todos os antibióticos ativos contra microrganismos Gram-positivos. Além disso, foi resistente à simulação do suco gástrico em pH 3, pancreatina e bile. O sobrenadante livre de células de L. lactis GU967439 inibiu o crescimento de Staphylococcus aureus ATCC 6538, Staphylococcus aureus ATCC 29213, Enterococcus faecalis ATCC 29212, Listeria innocua ATCC 33090, Streptococcus bovis 2.5 (WT) e mostrou melhor atividade do que a cepa N° 11454 de L. lactis ATCC, no entanto, nenhuma das cepas avaliadas apresentou atividade contra os microrganismos Gram-negativos (Escherichia coli ATCC 25922, Pseudomonas aeruginosa ATCC 9027). Esses resultados abrem caminho para estudos futuros que nos permitirão continuar a caracterizar o potencial probiótico de L. lactis GU967439.
Palavras-chave: bactérias lácticas, resistência gastrointestinal, atividade antimicrobiana, bacteriocinas, resistência a antibióticos.
Introducción
Los probióticos son microorganismos vivos que, al ser ingeridos en cantidad suficiente, ejercen efectos benéficos en la salud del hospedador. Para seleccionar estas cepas es necesario determinar su seguridad, características fisiológicas y efectos benéficos in vitro previos a la realización de ensayos in vivo (FAO y WHO, 2002). En relación con la seguridad de las bacterias candidatas a probióticos, uno de los principales criterios evaluados es la ausencia de resistencias adquiridas transferibles a antibióticos (Vizoso-Pinto et al., 2006).
La selección de bacterias nativas con propiedades tecnológicas y probióticas es fundamental para el desarrollo de productos lácteos funcionales. Desde el punto de vista tecnológico, las bacterias autóctonas (o nativas) presentan una fase de latencia de crecimiento más corta y son más eficientes acidificando la matriz, siendo de elección para la producción de alimentos fermentados (Torres et al., 2019). Los probióticos deben resistir a la presencia de enzimas en la cavidad oral, al pH ácido en el estómago y a la presencia de bilis en el duodeno (Kimoto-Nira et al., 2010) para llegar viables y en cantidad suficiente al intestino grueso, donde la mayoría de estos microorganismos desempeña su rol benéfico (Mainville et al., 2005). Entre los beneficios asociados a los probióticos se destaca el control de patógenos intestinales por la producción de compuestos antimicrobianos, entre ellos: ácido láctico y acético, peróxido de hidrógeno y bacteriocinas (péptidos con capacidad antimicrobiana) (Stoyanova et al., 2012).
Las bacterias probióticas más utilizadas son Lactobacillus spp., un género de bacterias ácidolácticas (BAL) y bifidobacterias. Las BAL se encuentran en ambientes muy nutritivos como la leche, la carne o los vegetales, y son los microorganismos más utilizados en el procesamiento de alimentos fermentados (Atanassova et al., 2003, Hladíková et al., 2012). Muchas investigaciones se han enfocado tanto en optimizar la participación de las BAL durante los procesos de elaboración como en ampliar sus aplicaciones utilizándolas como probióticos.
Lactococcus lactis, una de las BAL utilizadas en la elaboración de alimentos lácteos fermentados, posee actividad antimicrobiana y se ha sugerido su uso en la producción de alimentos funcionales (Zhou et al., 2010). Anteriormente, algunos autores comprobaron la actividad antilisterial in vitro y en quesos de L. lactis GU967439 aislada de leche cruda proveniente de una quesería artesanal de Nueva Helvecia, Colonia, Uruguay (Fraga, et al., 2013, González-Revello et al., 2016). Para continuar caracterizando la cepa, el presente trabajo se propuso evaluar la resistencia a antibióticos, el efecto del pasaje por el tracto gastrointestinal y la actividad antimicrobiana in vitro de la cepa nativa de L. lactis GU967439.
Materiales y Métodos
Cepas bacterianas
Se utilizó L. lactis GU967439, aislada de leche cruda proveniente de una quesería artesanal de Nueva Helvecia, Colonia, Uruguay, y la cepa de referencia American Type Culture Collection (ATCC) Nº 11454 de L. lactis.
Sensibilidad a antibióticos
Se determinó la sensibilidad a antibióticos siguiendo el método de difusión de Kirby–Bauer con modificaciones propuestas por Ture y Boran (2015). Se suspendieron colonias aisladas de cultivos frescos de L. lactis en diluyente estéril (NaCl 0,85 %) para lograr una turbidez similar a 0,5 de la escala Mc Farland (1,5 x 108 UFC/mL aproximadamente). Las suspensiones fueron inoculadas con hisopos estériles en placas que contenían Müller-Hinton agar (HiMedia®, India). Posteriormente, se depositaron discos con antibiótico (Oxoid Ltd., Basingstoke, UK) utilizando pinzas estériles, y se incubaron en estufa a 30 ℃ en aerobiosis, durante 24 horas. Los discos utilizados contenían uno de los siguientes antibióticos (microgramos/disco): penicilina (10 U), ampicilina (10 µg), piperacilina (100 µg), carbenicilina (100 µg), amoxicilina y clavulánico (30 µg), cefalotina (30 µg), cefradina (30 µg), cefuroxima (30 µg), cefepima (30 µg), cefotaxima (30 µg), ceftazidima (30 µg), cefoperazona (75 µg), imipenem (10 µg), aztreonam (30 µg), teicoplanina (30 µg), amicacina (30 µg), gentamicina (120 µg), kanamicina (30 µg), neomicina (30 µg), estreptomicina (10 µg), tobramicina (10 µg), telitromicina (15 µg), doxiciclina (30 µg), linezolid (30 µg), clindamicina (2 µg), cloranfenicol (30µg), ácido nalidixílico (30 µg), moxifloxacina (5 µg), levofloxacina (5 µg), pefloxacina (5 µg), colistin (10 µg), eritromicina (5 µg), vancomicina (5 µg) y tetraciclina (30 µg).
La lectura se realizó midiendo los halos de inhibición en milímetros, incluyendo el diámetro de los discos de antibióticos, y se clasificaron en resistentes, moderadamente sensibles y sensibles, según Charteris et al. (1998a), Clinical & Laboratory Standards Institute (CLSI, 2009), Cetinkaya et al. (2012), Khemariya et al. (2013), Haghshenas et al. (2014), y Ture y Boran (2015).
Resistencia a la simulación del pasaje por el tracto gastrointestinal
Resistencia a jugos gástricos y pancreáticos
La tolerancia a los jugos gástricos y pancreáticos se testeó de acuerdo a la metodología propuesta por Charteris et al. (1998b), con algunas modificaciones (Monteagudo-Mera et al., 2012). Se simuló el jugo gástrico suspendiendo pepsina porcina (Sigma, USA) en solución fisiológica salina (NaCl, 0,5 %, p/v) a una concentración final de 3 g/L y se ajustó el pH a 2, 2,5 y 3 con HCl 5M. El jugo pancreático se preparó suspendiendo pancreatina en una solución salina estéril (0.5 % NaCl, p/v) a una concentración final de 1 g/L y ajustando el pH a 8,0 con NaOH 5M.
Se obtuvieron cultivos overnight (concentración aproximada de 1 x 108 UFC/mL) de L. lactis en caldo MRS (HiMedia®, India), y se tomaron alícuotas de 1,0 mL en tubos tipo eppendorf estériles, que fueron centrifugados (10000 RPM, 10 minutos, 4 ℃) y lavados 2 veces con PBS (10 Mm, pH 7). Los pellets de células fueron resuspendidos en 0,3 mL de NaCl (0,5 % p/v) y adicionados con 1 mL de solución gástrica o pancreática, y luego incubados en baño María a 37 ℃ durante 120 y 240 minutos respectivamente.
Se realizaron recuentos de células viables en MRS agar (HiMedia®, India) de los cultivos overnight y posterior a la incubación. Finalmente, se determinó la tasa de supervivencia de L. lactis luego de la incubación a pH 2, 2,5 y 3 de acuerdo a la siguiente ecuación:
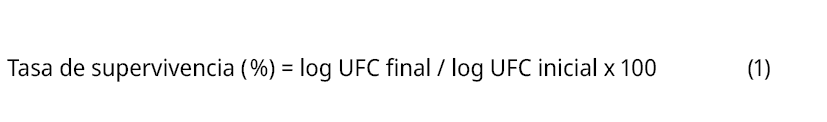
Tolerancia a las sales biliares
Para evaluar la tolerancia a las sales biliares se utilizó el método propuesto por Maragkoudakis et al. (2006). Se obtuvieron cultivos overnight que fueron centrifugados y lavados de la misma manera que para el ensayo anterior, y luego resuspendidos en 5 mL de caldo MRS (HiMedia®, India) suplementado con bilis bovina al 0,3 % para obtener una concentración aproximada de 2 x 107 UFC/mL. La suspensión se incubó a baño María a 37 ℃ por 240 minutos. Se tomaron alícuotas (0,1 mL) de cada cultivo previo a la incubación y luego de la misma para realizar recuento de células viables en MRS agar (HiMedia®, India), y se calculó la tasa de supervivencia.
Actividad antimicrobiana
La capacidad antimicrobiana se evaluó mediante el método Agar well diffusion (Fraga, 2008) frente a los siguientes microorganismos: Staphylococcus aureus ATCC 6538, Staphylococcus aureus ATCC 29213, Enterococcus faecalis ATCC 29212, Listeria innocua ATCC 33090, Streptococcus bovis 2.5 (WT), Escherichia coli ATCC 25922 y Pseudomonas aeruginosa ATCC 9027.
Para ello, se utilizaron 25 mL de caldo Brain Heart Infusion (BHI, HiMedia®, India) para S. aureus, E. coli, E. faecalis y P. auriginosas, caldo BHI suplementado con 0,6 % (p/v) de extracto de levadura para L. innocua y caldo MRS para S. bovis, adicionados de agar al 0,8 %. Los medios fundidos y atemperados a 45 ℃ fueron inoculados con cultivos overnight de los microorganismos indicadores, obteniendo una concentración final aproximada de 106 UFC/mL y dispuestos en placas de Petri que contenían cilindros metálicos de 10 mm. Una vez solidificado el medio de cultivo, se retiraron los cilindros quedando en su lugar pocillos que fueron inoculados con 100 µl de diluciones seriadas (1⁄2, 1⁄4, 1⁄8, 1⁄16, 1⁄32, etc.), con solución buffer fosfato estéril (PBS, 10 Mm pH 7) del sobrenadante libre de células (SLC) de L. lactis.
El SLC se obtuvo de la siguiente manera: a partir de un cultivo overnight de L. lactis en caldo MRS, se inoculó un matraz que contenía 100 mL de caldo MRS para obtener una concentración aproximada de 1 x 103 UFC/mL y se incubó a 30 ℃ durante 11 horas en aerobiosis. Se determinó el recuento de L. lactis tras la incubación mediante siembra de gotas en superficie en MRS agar, y se incubaron a 30 ℃, durante 24 horas en microaerofilia. Posteriormente, se obtuvieron alícuotas de 1,5 mL en tubos tipo eppendorf estériles y fueron tratados con ondas de ultrasonido (Jeken®, Digital Ultrasonic Cleaner PS-06ª, 50W, 40 kHz, 160 segundos) para favorecer la liberación de bacteriocinas adsorbidas a las células productoras (Rodríguez, 2016). Por último, el SLC se obtuvo centrifugando los cultivos a 10000 RPM durante 15 minutos a 4 ℃ y se ajustó a pH 6 con NaOH 1M para eliminar el efecto inhibitorio de los ácidos orgánicos. Se utilizó 100 μL de Nisina (Nisaplin®, Danisco) en una concentración de 100 mg/mL como control positivo y 100 μL de MRS ajustado a pH 6 con NaOH 1M como control negativo. Las placas fueron incubadas en aerobiosis a 37 ℃ durante 24 horas y la producción de bacteriocinas se expresó en unidades arbitrarias de actividad por mililitro (UA/mL), definidas como el inverso de la dilución más alta que presentó halo de inhibición de crecimiento (Schillinger y Lucke, 1991).
Análisis estadísticos
La resistencia al jugo gástrico a pH 2, 2,5 y 3 se analizó mediante el test de Student de 2 colas y la resistencia a la pancreatina y bilis se analizó utilizando el test de Welch. La actividad antimicrobiana de L. lactis GU967439 y ATCC Nº 11454 se comparó mediante el test de Mann Whitney. Los análisis fueron realizados utilizando el software Rstudio (versión 4.3.2). Para todos los casos se utilizó α = 0.05, y los ensayos se realizaron por triplicado de forma independiente.
Resultados
Sensibilidad a antibióticos
L. lactis ATCC N° 11454 y GU967439 fueron sensibles a todos los antibióticos activos frente a microorganismos Gram positivos y fueron resistentes al ácido nalidixílico, colistina, estreptomicina y aztreonam.
Resistencia a la simulación del pasaje por el tracto gastrointestinal
En la Tabla 1 se observa el efecto de la simulación de los jugos gástricos a distintos valores de pH (3, 2,5 y 2) en el recuento de células viables (expresado como log10UFC), la tasa supervivencia y la comparación entre los recuentos iniciales y finales tras 120 minutos de incubación de las cepas de L. lactis.
Tabla 1. Supervivencia de L. lactis a la simulación del jugo gástrico a pH 3, 2,5 y 2 durante 120 min (n = 3, ± DE).
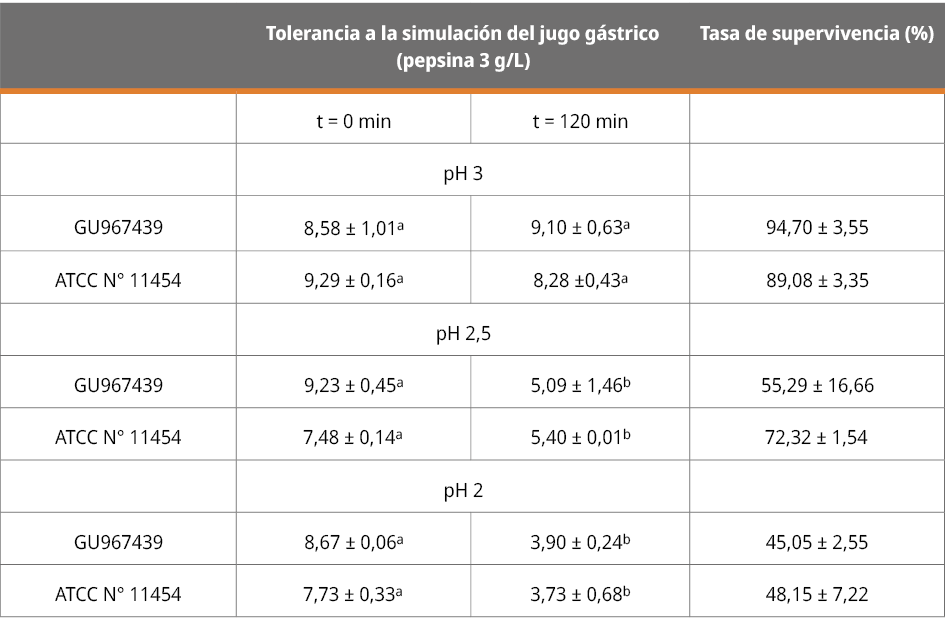
Valores representan la media de tres réplicas. ± Desvío estándar de la media. Valores con superíndices distintos indican diferencias significativas (p < 0.05, test de Welch).
Las cepas presentaron una reducción significativa de células viables en la simulación del jugo gástrico a valores de pH de 2 y 2,5; sin embargo, ambas mostraron células viables.
Tabla 2. Supervivencia de L. lactis a la simulación del jugo pancreático (pancreatina 1 g/L, pH = 8) durante 240 minutos (n = 3, ± DE).
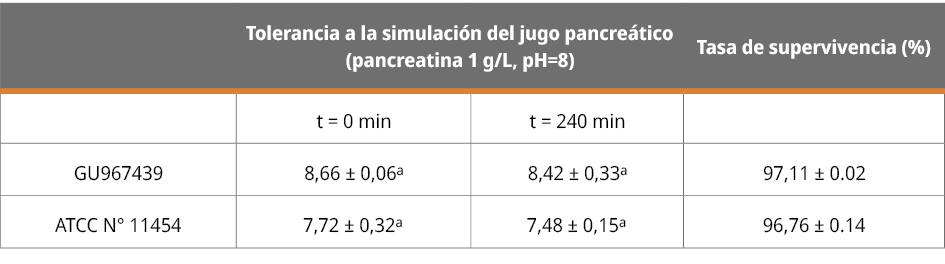
Valores representan la media de tres réplicas. ± Desvío estándar de la media. Valores con superíndices distintos indican diferencias significativas (p < 0.05, test de Student de 2 colas).
Tabla 3. Supervivencia de L. lactis a la bilis bovina (0.3 %) durante 240 minutos (n = 3, ± DE).

Valores representan la media de tres réplicas. ± Desvío estándar de la media. Valores con superíndices distintos indican diferencias significativas (p<0.05, test de Student de 2 colas).
Finalmente, no se observaron diferencias significativas (p > 0.05) en el recuento de células viables tras la incubación de L. lactis en presencia de pancreatina o bilis.
Actividad antimicrobiana
En la Tabla 4 se muestra la actividad inhibitoria del SLC de las cepas de L. lactis expresada como medianas de UA/mL frente a cada microorganismo indicador, y la comparación de la actividad entre las cepas de L. lactis.
Tabla 4. Actividad antimicrobiana del SLC en unidades arbitrarias de actividad por mililitro (UA/mL) de L. lactis frente a microorganismos indicadores.
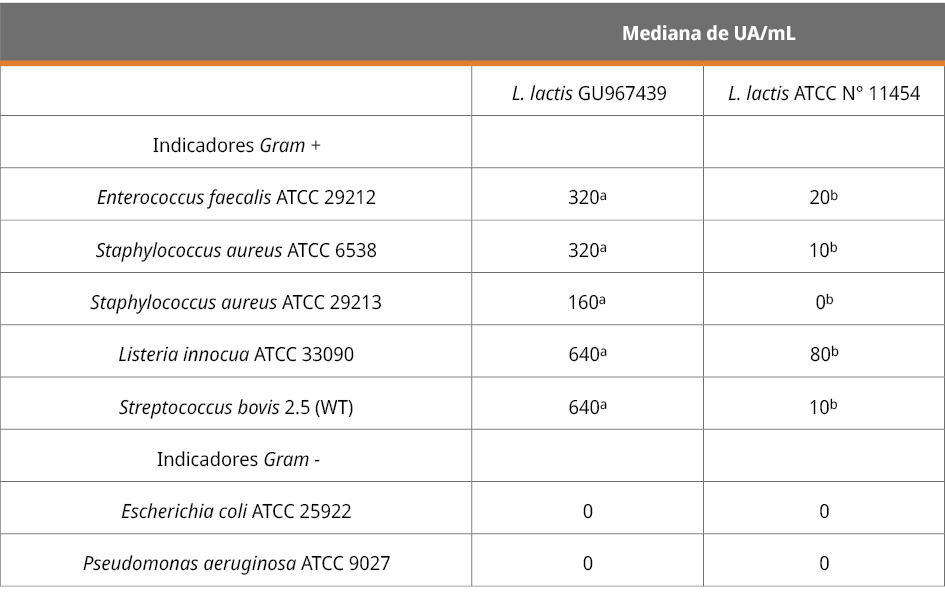
Valores con superíndices distintos indican diferencias significativas ( p < 0.05, test de Mann Whitney).
El SLC de L. lactis GU967439 fue capaz de inhibir todos los microorganismos Gram positivos utilizados como indicadores y mostró mejor actividad que el de L. lactis ATCC N° 11454 (p < 0.05). Por otro lado, ninguna de las cepas presentó actividad frente a los microorganismos Gram negativos utilizados como indicadores.

Figura 1. Resultado de la actividad antimicrobiana expresada en unidades arbitrarias de actividad por mililitro (UA/mL) con halos de inhibición del SLC de Lactococcus lactis GU967439 frente a Staphylococcus aureus ATCC N° 538.
Discusión
Sensibilidad a antibióticos
Las bacterias multirresistentes constituyen una amenaza emergente para el sistema alimentario. Las bacterias probióticas pueden participar en la transferencia de genes resistentes a antibióticos a la microbiota patógena o comensal del intestino, lo que incrementa la necesidad de evaluar la seguridad de los microorganismos utilizados (Selvin et al., 2020). La expresión del mismo patrón de sensibilidad en L. lactis GU967439 y ATCC 11454 sugiere que L. lactis GU967439 no adquirió elementos de resistencia a antibióticos.
Resistencia a la simulación del pasaje por el tracto gastrointestinal
Las cepas bacterianas probióticas deben sobrevivir a condiciones desfavorables durante su pasaje por el tracto gastrointestinal para ser capaces de colonizar el colon y ejercer un efecto benéfico en la salud del consumidor/huésped (Dimitrellou et al., 2016). Particularmente, la resistencia al pH bajo (pH 1,5 - 3,0) del estómago es uno de los criterios más importantes en la selección de probióticos (Solieri et al., 2014). Narimani et al. (2015) clasificaron 144 cepas en 4 grupos de acuerdo con la tasa de supervivencia a pH 2,5 por tres horas en susceptible (Tasa de supervivencia <10 %), moderadamente resistente (Tasa de supervivencia 10 - 60 %), resistencia buena (Tasa de supervivencia 60 - 80 %) y resistencia muy buena (Tasa de supervivencia >80 %). De acuerdo a la clasificación mencionada, L. lactis GU967439 es clasificado como muy resistente a pH3 (Tasa de supervivencia 94,7 %) y moderadamente resistente a pH 2,5 y 2 (Tasa de supervivencia 55,19 % y 45,05 % respectivamente). Sin embargo, no se conoce con exactitud si la resistencia a la simulación de las condiciones gástricas infiere de forma efectiva la tolerancia al ambiente gástrico in vivo.
Los alimentos funcionan como vehículos protectores de las cepas probióticas. Específicamente, los quesos presentan características que favorecen la viabilidad de estos microorganismos. Entre ellas se encuentran la disponibilidad de nutrientes, la formación de una matriz sólida de grasa y proteína con alta capacidad buffer, un bajo contenido de oxígeno y pH alto en comparación a otros productos lácteos (Rolim et al., 2020). Por lo anterior, posiblemente la viabilidad de L. lactis GU967439 se incremente aún más al ser administrado como parte de productos lácteos.
Asimismo, la habilidad de tolerar las enzimas pancreáticas presentes en el intestino delgado es otro criterio de selección de bacterias probióticas (Padmavathi et al., 2018). En el presente estudio, la presencia de pancreatina no redujo significativamente el recuento de L. lactis GU967439, por lo que no representaría una barrera para su viabilidad. Otro factor estresante al que se exponen los probióticos son las sales biliares que desorganizan las membranas celulares bacterianas (Ruiz et al., 2013). Tras la incubación durante 240 minutos de L. lactis GU967439 con bilis al 0,3 %, no se encontraron diferencias significativas entre los recuentos iniciales y finales.
Actividad antimicrobiana
Lorenzo y Raffo (2015) demostraron la producción de diacetilo por L. lactis GU967439. Posteriormente, Rodríguez (2016) caracterizó el SLC y concluyó que la bacteriocina contenida posiblemente se trate de un lantibiótico distinto a la nisina. Más adelante, Hernández (2021) detectó mediante PCR la presencia del gen de la nisina en el genoma de L. lactis GU967439 y a su vez, el espectro de masas reveló una señal similar a la de la nisina Z. Por lo anterior, la mejor actividad antimicrobiana de L. lactis GU967439 es posible que se deba a que produce más de un compuesto con actividad antimicrobiana. Por otro lado, las bacteriocinas presentan una elevada actividad antimicrobiana frente a cepas relacionadas (Yongkiettrakul et al., 2019), lo que explicaría la ausencia de actividad frente a microorganismos Gram negativos testeados en este estudio.
Conclusiones
La demanda creciente de nuevos alimentos funcionales lleva a la necesidad de caracterizar y seleccionar nuevas cepas probióticas. En el presente trabajo se demostró que L. lactis GU967439 posiblemente no adquirió elementos de resistencia a antibióticos de otras cepas, representando una cepa segura para su consumo. Además, presentó una tasa de supervivencia adecuada a la simulación del jugo gástrico a pH 3, y resistió la presencia de pancreatina y bilis. Asimismo, el SLC de L. lactis GU967439 exhibió actividad inhibitoria in vitro frente a un amplio rango de microorganismos Gram positivos utilizados como indicadores y presentó mejor actividad que la cepa L. lactis ATCC N° 11454. Sin embargo, ni L. lactis ATCC N° 11454 ni L. lactis GU967439 presentaron actividad frente a los microorganismos Gram negativos empleados. Estos resultados dan lugar a futuros trabajos que permitan continuar caracterizando el potencial probiótico de L. lactis GU967439.
Agradecimientos
A la Comisión de Investigación y Desarrollo Científico (CIDEC), de la Facultad de Veterinaria, de la Universidad de la República.
Referencias
Atanassova, M.; Choiset, Y.; Dalgalarrondo, M.; Chobert, J.; Dousset, X.; Ivanova, I. y Haertle, T., 2003. Isolation and partial biochemical characterization of a proteinaceous anti-bacteria and anti-yeast compound produced by Lactobacillus paracasei subsp. paracasei strain M3. En: International Journal of Food Microbiology, (87), pp. 63-73. DOI: https://doi.org/10.1016/S0168-1605(03)00054-0
Cetinkaya, F.; Coplu, C.; Simsek, H.; Mus, T.T. y Cibik, R., 2012. Antibiotic susceptibility of Lactococcus isolated from Turkish raw milk cheeses. En: Medycyna Wet, (68), pp. 49-53.
Charteris, W. P.; Kelly, P. M.; Morelli, L. y Collins, J. K., 1998a. Antibiotic susceptibility of potentially probiotic Lactobacillus species. En: Journal of food protection, (61), pp. 1636-1643. DOI: https://doi.org/10.4315/0362-028X-61.12.1636
Charteris, W.P.; Kelly, P.M.; Morelli, L. y Collins, J.K.,1998b. Development and application of an in vitro methodology to determine the transit tolerance of potentially probiotic Lactobacillus and Bifidobacterium species in the upper human gastrointestinal tract. En: Journal of applied microbiology, (84), pp. 759-768. DOI: https://doi.org/10.1046/j.1365-2672.1998.00407.x
Clinical & Laboratory Standards Institute (CLSI), 2009. Performance standards for antimicrobial susceptibility testing. 19th International Supplement, M100–MS19. Pittsburgh: CLSI
Dimitrellou, D.; Kandylis, P.; Petrovic, T.; Dimitrijevic-Brankovic, S.I.; Levic, S.; Nedovic, V. y Kourkoutas, Y., 2016. Survival of spray dried microencapsulated Lactobacillus casei ATCC 393 in simulated gastrointestinal conditions and fermented milk. En: LWT Food Science and Technology,(71), pp. 169–174. DOI: https://doi.org/10.1016/j.lwt.2016.03.007
FAO y WHO, 2002. Guidelines for the evaluation of probiotics in food. Ontario: FAO y WHO.
Fraga, M., 2008. Vaginal lactic acid bacteria in the mare: evaluation of the probiotic potential of native Lactobacillus spp. and Enterococcus spp. strains. En: Antonie van Leeuwenhoek; (93), pp. 71-78. DOI: https://doi.org/10.1007/s10482-007-9180-4
Fraga, M.; Perelmuter, K.; Giacaman, S. S.; Zunino, P. M. y Carro, S. B., 2013. Antimicrobial properties of lactic acid bacteria isolated from Uruguayan artisan cheese. En: Food Science and Technology, (33), pp. 801-804. DOI: https://doi.org/10.1590/S0101-20612013000400029
González - Revello, Á.; Carro, S.; Cal, K.; Giacaman, S. y Aldrovandi, A., 2016. Lactococcus lactis autóctono: evaluación del efecto antilisterial y de propiedades sensoriales en quesos tipo Cuartirolo. En: Innotec, (12), pp. 15-26. DOI: https://doi.org/10.26461/12.02
Haghshenas, B.; Abdullah, N.; Nami, Y.; Radiah, D.; Roseli, R. y Khosroushahi, Y., 2014. Different effects of two newly-isolated probiotic Lactobacillus plantarum 15HN and Lactococcus lactis subsp. Lactis 44Lac strains from traditional dairy products on cancer cell lines. En: Anaerobe, (30), pp. 51-59. DOI: https://doi.org/10.1016/j.anaerobe.2014.08.009
Hernández, M., 2021. Identificación y caracterización de una sustancia inhibidora tipo bacteriocina producida por una cepa nativa de Lactococcus lactis aislada de queserías artesanales. Montevideo: Universidad de la República. Facultad de Ciencias. (Tesis de Grado).
Hladíková, Z.; Smetankova, J.; Greif, G. y Greifova, M., 2012. Antimicrobial activity of selected lactic acid cocci and production of organic acids. En: Acta Chimica Slovaca, (5), pp. 80-85. DOI: https://doi.org/10.2478/v10188-012-0013-3
Kimoto-Nira, H.; Suzuki, C.; Sasaki, K.; Kobayashi, M. y Mizumachi, K., 2010. Survival of a Lactococcus lactis strain varies with its carbohydrate preference under in vitro conditions simulated gastrointestinal tract. En: International Journal of Food Microbiology, (143), pp. 226–229. DOI: https://doi.org/10.1016/j.ijfoodmicro.2010.07.033
Khemariya, P.; Singh, S.; Nath, G. y Gulati, A. K., 2013. Isolation, identification, and antibiotic susceptibility of nisC Lactococcus lactis from dairy and non-dairy sources. En: Czech Journal of Food Sciences, (31), pp. 323–331. DOI: https://doi.org/10.17221/316/2012-CJFS
Lorenzo, G. y Raffo, M., 2015. Lactococcus lactis nativo: caracterización de la producción de bacteriocinas, propiedades tecnológicas y efecto antimicrobiano sobre Listeria innocua. Montevideo: Universidad de la República. Facultad de Veterinaria. (Tesis de Grado).
Mainville, I.; Arcand, Y. y Farnworth, E. R., 2005. A dynamic model that simulates the human upper gastrointestinal tract for the study of probiotics. En: International Journal of Food Microbiology, (99), pp. 287-296. DOI: https://doi.org/10.1016/j.ijfoodmicro.2004.08.020
Maragkoudakis, P. A.; Zoumpopoulou, G.; Miaris, C.; Kalantzopoulos, G.; Pot, B. y Tsakalidou, E., 2006. Probiotic potential of Lactobacillus strains isolated from dairy products. En: International Dairy Journal, 16, pp. 189–199. DOI: https://doi.org/10.1016/j.idairyj.2005.02.009
Monteagudo-Mera, A.; Rodríguez-Aparicio, L.; Rúa, J.; Martínez- Blanco, H.; Navasa, N. y García-Armesto, M. R., 2012. In vitro evaluation of physiological probiotic properties of different lactic acid bacteria strains of dairy and human origin. En: Journal of Functional Foods, (4), pp. 531–541. DOI: https://doi.org/10.1016/j.jff.2012.02.014
Narimani, T.; Tarinejad, A. y Hejazi, M. A., 2015. Isolation and biochemical and molecular identification of Lactobacillus bacteria with probiotic potential from traditional cow milk and yogurt of Khoi city. En: Journal of Food Science & Technology, 12(48).
Padmavathi, T.; Bhargavi, R.; Priyanka, P. R.; Niranjan, N. R. y Pavitra, P. V., 2018. Screening of potential probiotic lactic acid bacteria and production of amylase and its partial purification. En: Journal of Genetic Engineering and Biotechnology, (16), pp. 357-362. DOI: https://doi.org/10.1016/j.jgeb.2018.03.005
Rodríguez, I., 2016. Implementación de una técnica de purificación de la/s bacteriocina/s producidas por Lactococcus lactis nativo. Montevideo: Universidad de la República. Facultad de Veterinaria. (Tesis de Grado).
Rolim, F. R.; Neto, O. C. F.; Oliveira, M. E. G.; Oliveira, C. J. y Queiroga, R. C., 2020. Cheeses as food matrixes for probiotics: in vitro and in vivo tests. En: Trends in Food Science & Technology, (100), pp. 138-154. DOI: https://doi.org/10.1016/j.tifs.2020.04.008
Ruiz, L.; Margolles, A. y Sánchez, B., 2013. Bile resistance mechanisms in Lactobacillus and Bifidobacterium. En: Frontiers in Microbiology, (4), pp. 396. DOI: https://doi.org/10.3389/fmicb.2013.00396
Selvin, J.; Maity, D.; Sajayan, A. y Kiran, G. S., 2020. Revealing antibiotic resistance in therapeutic and dietary probiotic supplements. En: Journal of Global Antimicrobial Resistance, (22), pp. 202-205.
Schillinger, U. y Lucke, F. 1991 El empleo de bacterias lácticas como cultivos protectores en productos cárnicos. En: Fleischwirtsch, (1), pp. 35-40.
Solieri, L.; Bianchi, A.; Mottolese, G.; Lemmetti, F. y Giudici, P., 2014. Tailoring the probiotic potential of non-starter Lactobacillus strains from ripened Parmigiano Reggiano cheese by in vitro screening and principal component analysis. En: Food Microbiology, (38), pp. 240–249. DOI: https://doi.org/10.1016/j.fm.2013.10.003
Stoyanova, L. G.; Ustyugova, E. A. y Netrusov, A. I., 2012. Antibacterial metabolites of lactic acid bacteria: their diversity and properties. En: Applied Biochemistry and Microbiology, (48) pp. 229-243.
Torres, S.; Fabersani, E.; Marquez, A. y Gauffifin-Cano, P., 2019. Adipose tissue inflammation and metabolic syndrome. The proactive role of probiotics. En: European Journal of Nutrition, (58), pp. 27–43.
Ture, M. y Boran, H., 2015. Phenotypic and genotypic antimicrobial resistance of Lactococcus sp. strains isolated from rainbow trout (Oncorhynchus mykiss). En: Journal of Veterinary Research, (59) pp. 37-42. DOI: https://doi.org/10.1515/bvip-2015-0006
Vizoso-Pinto, M. G.; Franz, C. M.; Schillinger, U. y Holzapfel, W. H. 2006. Lactobacillus spp. with in vitro probiotic properties from human feces and traditional fermented products. En: International Journal of Food Microbiology, (109), pp.205–214. DOI: https://doi.org/10.1016/j.ijfoodmicro.2006.01.029
Yongkiettrakul, S.; Maneerat, K.; Arechanajan, B.; Malila, Y.; Srimanote P. y Gottschalk, M., 2019. Antimicrobial susceptibility of Streptococcus suis isolated from diseased pigs, asymptomatic pigs, and human patients. En: Thailand. BMC Veterinary Research, (15), pp. 1-12.
Zhou, X.; Wang, Y.; Yao, J. y Li, W., 2010. Inhibition ability of probiotic, Lactococcus lactis, against A. hydrophila and study of its immunostimulatory effect in tilapia (Oreochromis niloticus). En: International Journal of Engineering, Science And Technology, (2), pp. 73-80.


