Autores
- REY BENTOS, FABIANA (1); CAPDEVIELLE SOSA, FABIÁN (2)
-
(1) LATITUD, FUNDACIÓN LATU. MONTEVIDEO, URUGUAY
(2) UNIVERSIDAD TECNOLÓGICA DEL URUGUAY. DURAZNO, URUGUAY
El mercado de productos botánicos se incrementa a nivel mundial debido a la preferencia de los consumidores por los productos naturales. Esto hace que sean blanco de adulteración, lo cual es más común y difícil de detectar en los derivados industrializados. Es crítico contar con procedimientos confiables que permitan su autentificación. En este estudio se aplicaron herramientas moleculares basadas en ADN para la generación de información genómica aplicada con el fin de clasificar muestras de hierbas aromáticas y medicinales. Se aplicó la estrategia del Código de Barras de ADN (DNA Barcoding) utilizando muestras de tejido fresco de identidad taxonómica conocida (referencias) y muestras industrializadas. Los resultados obtenidos indican que los marcadores rbcL y matK son amplificables con los cebadores utilizados, obteniéndose secuencias bidireccionales de alta calidad, en el caso de tejido fresco en el 83% de los casos para rbcL y en el 63% de los casos para matK. En las muestras industrializadas los valores fueron de 50% y 35% respectivamente. La herramienta BOLD permitió la clasificación de la mayor parte de las muestras. Esta metodología es una herramienta útil y accesible que permite la clasificación en grupos, si bien su desempeño se ve acotado por la representación de las especies en las bases de datos.
PALABRAS CLAVES: barcodes rbcL y matK, hierbas aromáticas y medicinales, clasificación.
O mercado dos produtos botânicos está em crescimento a nível mundial, devido à preferência dos consumidores por produtos naturais. Isto torna-os alvos de adulteração, o que é mais comum e difícil de detectar nos derivados industrializados. É fundamental dispor de procedimentos fiáveis que permitam a sua autenticação. Neste estudo, foram aplicadas ferramentas moleculares baseadas no ADN para gerar informação genómica aplicada para classificar amostras de ervas aromáticas e medicinais frescas e industrializadas. O trabalho baseou-se na estratégia de codificação de barras de ADN utilizando amostras de tecido fresco de identidade taxonómica conhecida (referências) e amostras industrializadas. Os resultados obtidos indicam que os marcadores rbcL e matK são amplificáveis com os iniciadores utilizados, obtendo sequências bidireccionais de alta qualidade no caso do tecido fresco em 83% dos casos para rbcL e em 63% dos casos para matK. Nas amostras industrializadas, os valores foram de 50% e 35%, respectivamente. A ferramenta BOLD permitiu a classificação da maioria das amostras. Esta metodologia é uma ferramenta útil e acessível que permite a classificação em grupos, embora o seu desempenho seja limitado pela representação das espécies nas bases de dados.
PALAVRAS CHAVE: barcodes rbcL e matK, ervas aromáticas e medicinais, classificação.
Introducción
En las últimas décadas, los conceptos de calidad, autenticidad y trazabilidad han acaparado el interés de los consumidores, así como de la industria, los productores, los comerciantes y las agencias reguladoras. Los consumidores exigen alimentos frescos, sabrosos, nutritivos y seguros. Más aún, un número cada vez más grande se inclina por el consumo de alimentos funcionales que ofrecen beneficios específicos para la salud (Opara, 2003).
En este escenario, las plantas aromáticas y medicinales han despertado gran interés por sus propiedades y por la necesidad de la población de establecer hábitos de vida más saludables. Son requeridas para uso culinario, medicinal, en perfumería y cosmética, aromaterapia, para la elaboración de suplementos alimenticios y extractos para la producción de nuevas drogas. Para ello se utilizan distintas partes de la planta (semillas, frutos, raíces, tallos, corteza, rizoma de las flores) con el fin de explotar sus propiedades terapéuticas, así como sus sabores o aromas (EFSA Scientific Committee, 2009), (World Health Organization, 1999). Como consecuencia, ha aumentado la demanda y el comercio de este tipo de plantas y sus derivados a nivel mundial. Un reporte publicado por Persistence Market Research da cuenta de este aumento, destacando que el valor del mercado global de suplementos botánicos en el año 2017 fue estimado en 40.000 millones de dólares y se proyecta en más de 65.000 millones de dólares para el año 2025 (Persistence Market Research, 2017).
A pesar de esto, la regulación de este tipo de productos, tanto para consumo alimentario como medicinal, aún sigue siendo muy variable entre los países (Schilter, et al., 2003). La ausencia de una regulación clara y compartida entre los diversos países, así como la creciente demanda de productos de alta calidad derivados de este tipo de plantas, ha conducido a un aumento en los casos de adulteración, sustitución y fraude (Frigerio, et al., 2019). Según un estudio publicado por Ichim, un 27% de los productos herbales que se ofrecen en el mercado global se encuentran adulterados ya sea porque contienen sustitutos no declarados, contaminantes, especies de relleno; o bien porque ninguna de las especies está declarada (Ichim, 2019).
Los criterios de identidad y pureza juegan un rol preponderante en el aseguramiento de la calidad y reproducibilidad de los productos derivados de estas materias primas, lo cual contribuye a su seguridad y eficacia (Joshi, 2004; Coutinho Moraes, et al., 2015). Los productos industrializados, deshidratados y en polvo son más propicios para las adulteraciones ya que el adulterante es más difícil de distinguir visualmente. Para estos fines es crítico contar con procedimientos confiables que permitan la autentificación. La Organización Mundial de la Salud (OMS) hace hincapié en la importancia de las metodologías cualitativas y cuantitativas que permitan estandarizar los materiales vegetales mediante su caracterización botánica, composición química y actividad biológica (Nikam, et al., 2012). Los métodos tradicionalmente utilizados para identificar especies en materiales botánicos se basan en la búsqueda de similitudes a nivel macroscópico y/o microscópico con respecto a un material estándar, así como en propiedades fisicoquímicas de los mismos. Estas metodologías presentan limitaciones para su uso en productos procesados en los que los caracteres morfológicos no se han preservado, pueden ser afectadas por las condiciones de cultivo y/o recolección, y requieren un alto nivel de experticia (Liu, et al., 2019; Lu, et al., 2019).
Los métodos moleculares basados en ADN permiten obtener información confiable ya que la composición genética es única para cada individuo y no se ve afectada por la edad, las condiciones fisiológicas, ni los factores ambientales. Como los marcadores no son específicos de un tejido pueden detectarse en cualquier estadio del desarrollo del individuo (Heubl, 2010). El código de barras de ADN (DNA Barcoding) fue propuesto como una alternativa para identificar especies, basado en el uso de una pequeña región del genoma ubicada en una posición específica, que se encuentra presente en forma universal en todos los linajes y funciona como identificador único de cada especie (Hebert, et al., 2003). Inicialmente fue diseñado para su aplicación en animales, pero su uso fue extendido a plantas y hongos (Kress, 2017). En animales, el sistema se basa en la amplificación de la región 5’ del gen de la subunidad 1 de la enzima citocromo oxidasa (coxI), ubicado en el genoma mitocondrial. Esta región, denominada CO1, cumple con los criterios establecidos para una región estándar (Kress y Erickson, 2007). En plantas, la elección de la región a utilizar fue compleja. La baja tasa de sustitución del ADN mitocondrial en plantas imposibilitó el uso de la región CO1, tal como sucede en animales. En 2009, luego de que se llevaran a cabo numerosos estudios, el Consortium for the Barcode of Life (CBOL) recomendó la combinación de rbcL y matK como regiones estándar para plantas terrestres (CBOL Plant Working Group, 2009). Ambas corresponden a porciones de genes funcionales ubicadas en el cloroplasto (Fazekas, et al., 2008). A partir de allí varios autores han sugerido la combinación de estos dos marcadores con otros para mejorar la discriminación, como el marcador nuclear ITS, el espaciador intergénico trnH-psbA o el intrón trnL del cloroplasto (Chen, et al., 2010; Taberlet, et al., 2007; Newmaster, et al., 2007; Fazekas, et al., 2008).
En los últimos años se ha observado un aumento significativo en el número de publicaciones referidas al uso del Código de Barras de ADN (DNA Barcoding) para identificar especies vegetales de uso culinario o medicinal, y para autentificar o trazar plantas y productos botánicos. Este hecho es de suma importancia ya que demuestra el interés de la comunidad científica por un método con estas características (Bruni, et al., 2010; Stoeckle, et al., 2011; De Mattia, et al., 2011; Kool, et al., 2012; Newmaster, et al., 2013; Galimberti, et al., 2014; Vassou, et al., 2015; Frigerio, et al., 2019; Stallman, et al., 2019; Lu, et al., 2019; Veldman, et al., 2020). Asimismo, algunos estudios se han enfocado en la búsqueda de contaminaciones, sustituciones y uso de especies más económicas con la finalidad de adulterar productos botánicos (Stoeckle, et al., 2011; Newmaster, et al., 2013).
El objetivo de nuestro trabajo fue evaluar y demostrar la aplicabilidad de herramientas moleculares basadas en la estrategia del Código de Barras de ADN (DNA Barcoding), para la clasificación de muestras de especies vegetales -frescas y procesadas- de interés industrial en nuestro país.
Materiales y Métodos
Material vegetal
Las especies incluidas en este trabajo se describen en la Tabla 1. Se utilizaron tres ejemplares de cada especie, excepto Origanum majorana y Petroselinum crispum, de las cuales se obtuvo una sola muestra. Los ejemplares fueron proporcionados por el Instituto Nacional de Investigación Agropecuaria (INIA), de su colección de germoplasma vinculada al proyecto FPTA N° 137 Desarrollo del sector de las plantas medicinales y aromáticas en Uruguay (Davies, 2004). Fueron usados como muestras de referencia por tener identificación taxonómica.
Además, se incluyeron muestras comerciales de algunas de las especies en estudio con el objetivo de evaluar la metodología en muestras problema. Las mismas fueron proporcionadas por el Departamento de Análisis de Productos Agropecuarios del LATU y por una empresa del ramo (Tabla 2).
Tabla 1. Descripción taxonómica de los materiales vegetales de referencia utilizados.
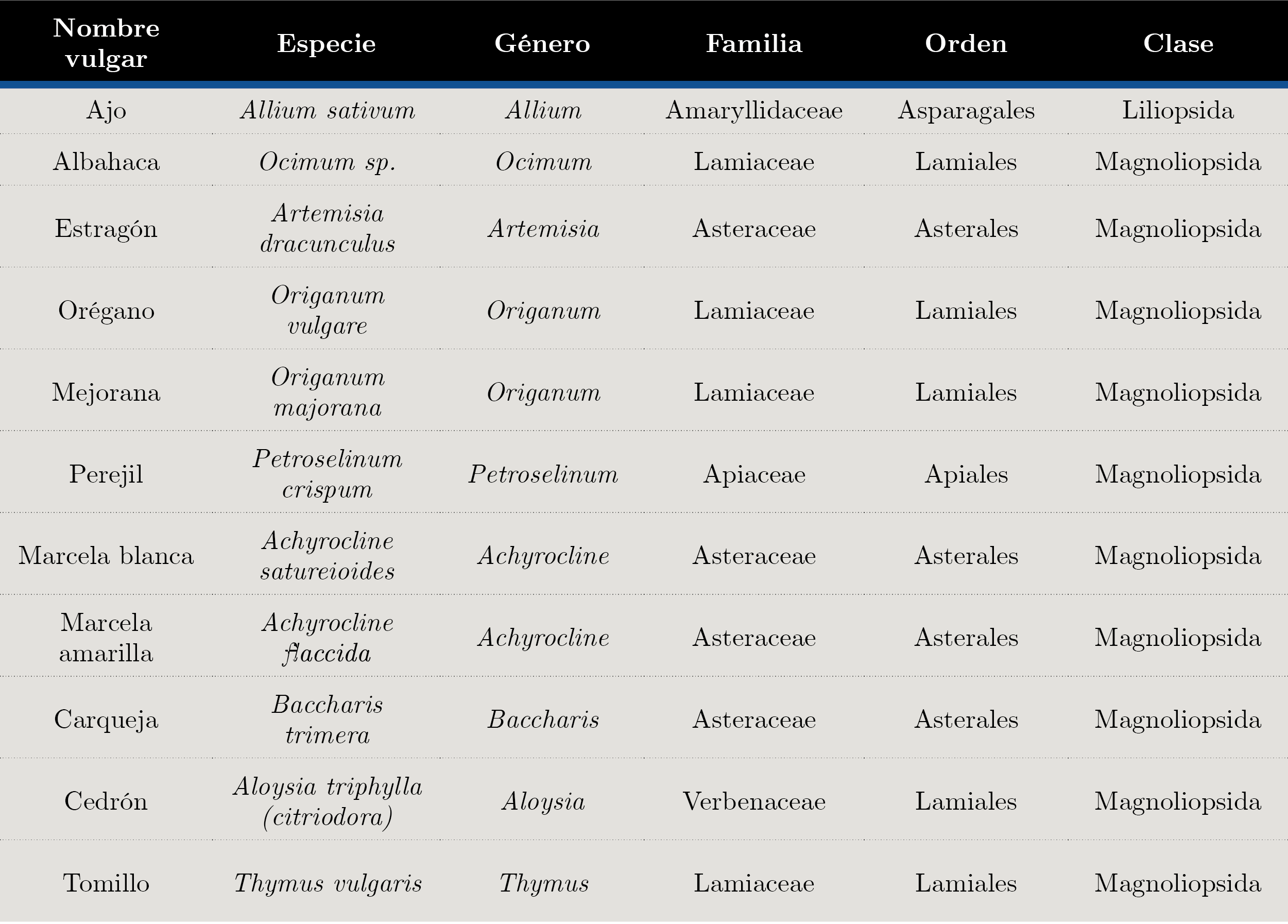
Tabla 2. Descripción de las muestras industrializadas comerciales analizadas.

Extracción de ADN
El tejido vegetal fue congelado con nitrógeno líquido y molido en mortero. Se tomó entre 0,5 y 1 g, se colocó en un tubo y se agregaron 600 μl de buffer de extracción (CTAB 2%, NaCl 1.4 M, EDTA 20 mM, Tris-Cl 100 mM pH 8.0, PVP-40 2%) precalentado a 60ºC y 1,2 μl de β-mercaptoetanol. Las muestras se incubaron a 60ºC por 30 min, mezclando de vez en cuando. Se le agregaron 600 μl de mezcla cloroformo-alcohol isoamílico (24:1), mezclando cuidadosamente, y se centrifugó a 10.000 rpm por 20 min a temperatura ambiente. El sobrenadante fue transferido a otro tubo y se agregaron 500 μl de isopropanol frío, mezclando suavemente hasta lograr la precipitación de los ácidos nucleicos. Las muestras fueron centrifugadas a 10.000 rpm por 20 min. El sobrenadante fue descartado y el pellet fue lavado dos veces con 500 μl de etanol 70%, seguido de una centrifugación a 10.000 rpm por 5 min. Los pellets se dejaron secar al aire a temperatura ambiente y luego fueron resuspendidos en 100 μl de agua milliQ® estéril y conservados en freezer a -20°C.
En el caso de algunas muestras industrializadas de origen comercial en las que se obtuvo muy bajo rendimiento de ADN utilizando el protocolo descrito anteriormente, se utilizó el kit comercial Nucleospin Food (Macherey – NagelGmbH & Co. KG, Düren, Germany), según las instrucciones del fabricante.
La evaluación de la calidad del ADN obtenido fue realizada mediante electroforesis en gel de agarosa al 1% (w/v), y se cuantificó en Nanodrop 1000 (Thermo Fisher Scientific, Wilmington, DE, USA).
Amplificación y secuenciación
Se amplificaron las regiones cloroplásticas rbcL y matK utilizando los cebadores diseñados por Erickson y Kim respectivamente, y cuyas secuencias se describen en la Tabla 3 (CBOL Plant Working Group, 2009).
Tabla 3. Secuencias de los cebadores utilizados y tamaño esperado del amplicón generado.

Las reacciones de amplificación se llevaron a cabo en un termociclador Palm-Cycler CG1-96 (Corbett Research, Mortlake, Australia). Las condiciones de reacción para el marcador rbcL fueron las siguientes: se agregaron 0.5 μl de ADN molde (200 ng en promedio) en 20 μl de mezcla de reacción. La misma contenía buffer 1X (Tris-HCl 10 mM pH 8,3, KCl 50 mM), MgCl2 1.0 mM, 0.2 mM de cada dNTP, 0.5 μM de cada cebador, DMSO 2%, 0.1 μg/ μl de BSA y 1.0 U de Standard Taq Polimerasa (New England Biolabs Inc., Ipswich, MA, USA). Las condiciones de reacción fueron las siguientes: un ciclo de desnaturalización a 95°C por3 min; 33 ciclos de desnaturalización a 94°C por 30 seg, unión de los cebadores a 55°C por 30 seg y extensión a 72°C por 1 min; y una extensión final a 72°C por 10 min.
Para la amplificación del marcador matK se tomaron 0.5 μl de ADN molde (200 ng en promedio) en 20 μl de mezcla de reacción conteniendo buffer 1X (Tris-HCl 10 mM pH 8,3, KCl 50 mM), MgCl2 1.0mM, 0.2 mM de cada dNTP, 1.0 μM de cada cebador, DMSO 4%, 0.4 μg/ μl de BSA y 2.0U de Standard Taq Polimerasa (New England Biolabs Inc., Ipswich, MA, USA). Las condiciones de reacción fueron las siguientes: un ciclo de desnaturalización a 94°C por 1 min; 35 ciclos de desnaturalización a 94°C por 30 seg, unión de los cebadores a 52°C por 20 seg y extensión a 72°C por 50 seg; y una extensión final a 72°C por 5 min.
Los productos de PCR fueron sometidos a electroforesis en gel de agarosa al 1.5% en buffer TAE (Tris- Acetato- EDTA) y visualizados por tinción con bromuro de etidio.
La purificación de los productos de PCR y la secuenciación por Sanger fueron realizadas por Macrogen Inc. (Seoul, Korea). Se utilizó un secuenciador 3730xl DNA Analyzer (Life Technologies, Carlsbad, USA). Las secuencias fueron editadas y ensambladas usando el software Geneious v. 7.1 (http://www.geneious.com) (Kearse et al., 2012). Se verificó la asignación de bases y la calidad de estas. En base a esto se recortaron los extremos, eliminando las posiciones en que no fue posible asignar una base (N) y aquellas cuya calidad fue muy baja. Las secuencias directa y reversa editadas, correspondientes a cada ejemplar, fueron ensambladas para dar lugar a una secuencia consenso.
Análisis de datos
Con la secuencia consenso obtenida para cada muestra se realizó la clasificación. Para ello se empleó la plataforma BOLD (http://www.boldsystems.org), que utiliza toda la biblioteca de secuencias estándar (barcode) de proyectos públicos y privados depositadas en su base de datos. En el caso de plantas, el motor de identificación de BOLD utiliza los marcadores rbcL y matK por defecto como herramienta para la clasificación, y el algoritmo BLAST en vez del algoritmo interno de BOLD. Como resultado se obtiene una lista de las noventa y nueve coincidencias más cercanas, pero no la probabilidad de clasificación en un taxón. A cada coincidencia se le asigna un porcentaje de identidad, un score y un valor E. La clasificación mediante BOLD no puede realizarse con ambos marcadores al mismo tiempo, es decir que se obtienen tantas asignaciones como secuencias barcode se utilicen (Ratnasingham y Hebert, 2007; Ratnasingham y Hebert, 2011; Boldsystems, 2019). A partir del listado de coincidencias que arroja la herramienta de identificación de BOLD, se definió como coincidencia más cercana el espécimen con mayor porcentaje de identidad, tomando como límite de aceptación arbitrario un 99,5%. En la mayoría de los casos este correspondió a la secuencia con mayor score. En las búsquedas en las que se obtuvieron múltiples coincidencias con igual porcentaje de identidad, se reportó el espécimen más cercano a la especie de referencia cuando fue posible. A su vez se registró la coincidencia más cercana perteneciente al mismo género (VMCIG) y la más cercana perteneciente a otro género (VMCDG).
Las muestras de referencia fueron utilizadas para validar la identificación taxonómica. La secuencia se consideró correctamente asignada cuando la especie a la que pertenece obtuvo el mayor score entre todos los candidatos. Se consideró una falla en la asignación cuando la especie correcta obtuvo un score menor o igual a otras especies.
Para el análisis de los datos de secuencias y especímenes, y como forma de validar la identificación, se utilizó la herramienta Taxon ID Tree disponible en BOLD. Esta permite la construcción de dendogramas usando el algoritmo Neighbour Joining. Las secuencias fueron alineadas con MUSCLE y para calcular las distancias se utilizó el modelo Kimura 2 Parameter.
Resultados y Discusión
En este trabajo se evaluó la aplicabilidad de la metodología del Código de Barras de ADN (DNA Barcoding) para la clasificación de muestras de hierbas de uso medicinal y de la industria alimentaria. Se utilizaron los marcadores recomendados por el grupo de trabajo de plantas del CBOL, rbcL y matK. rbcL comprende una región de 599 pares de bases ubicada en la región 5’ del gen que codifica para la ribulosa bifosfato carboxilasa (RuBisCO), localizada entre las bases 1 - 599 en la secuencia del gen en Arabidopsis thaliana. (Mondal, et al., 2013). Es sencilla de amplificar, secuenciar y alinear en la mayoría de las plantas. A pesar de su bajo poder de discriminación, se la considera importante por tratarse de una región de referencia muy usada en estudios filogenéticos que permite reconstruir relaciones entre especímenes a nivel de familia y género (Kress y Erickson, 2007). La región matK comprende 841 bases en el centro del gen que codifica para la enzima maturasa K. Se encuentra ubicada entre las bases 205 y 1046 en la secuencia completa del gen en el genoma plastídico de A. thaliana. Su tasa de sustitución es tres veces más alta que la de rbcL a nivel de nucleótidos y seis veces mayor a nivel de aminoácidos, lo que muestra que es un gen de rápida evolución. A diferencia de rbcL, matK resulta más difícil de amplificar, particularmente en las plantas que no son angiospermas. Pero debido a su rápida evolución, es la más parecida a CO1 (la región estándar usada en animales), brindando un alto poder de discriminación en plantas (Vijayan, et al., 2010).
Con el protocolo de extracción de ADN puesto a punto, se pudo extraer ADN en cantidad considerable de todos los tejidos frescos (entre 114 y 2800 ng/µL), siendo la muestra de Ocimum sp. de la que se obtuvo menos cantidad (114 ng/µL). En todos casos la calidad del ADN extraído fue buena (A260/280 entre 1,4 y 3,2). En el caso de las muestras comerciales en las que los tejidos vegetales fueron sometidos a algún tipo de procesamiento (deshidratación, molienda, extracción), el rendimiento fue bueno (entre 40 y 2300 ng/µL) y también la calidad (A260/280 entre 1,4 y 2,3), excepto para las muestras de arándano en cápsulas y ajo en escamas. En estos dos casos se volvió a extraer el ADN usando un kit comercial basado en el uso de sales caotrópicas, agentes desnaturalizantes y detergentes en el buffer de lisis, y la adsorción del ADN a una membrana de silica, lo que permitió la remoción de impurezas mediante lavados de la membrana con buffer. Con este procedimiento se logró mejorar el rendimiento en ambas muestras y la pureza del ADN extraído del ajo en escamas, pero no del proveniente del arándano, lo cual tiene sentido por la alta concentración de polifenoles que contienen los ejemplares de esta especie en sus hojas y frutos.
Con respecto a la amplificación de la región rbcL, con el protocolo puesto a punto se analizaron 29 muestras de referencia (tejido fresco) pertenecientes a las familias Amaryllidaceae, Apiaceae, Astereaceae, Lamiaceae, Verbenaceae. En todos los casos se obtuvieron productos de amplificación del tamaño esperado. Fueron analizadas 9 muestras industrializadas de origen comercial de perejil, tomillo, albahaca, estragón, orégano, ajo en escamas, ajo molido, brócoli y arándano en las presentaciones que se detallan en la Tabla 2. En todos los casos se obtuvieron productos de amplificación, excepto a partir de la muestra de arándano que no amplificó. Este hecho era de esperar ya que en la extracción se obtuvo poco ADN y de baja pureza. De acuerdo con los resultados obtenidos podemos corroborar que los cebadores rbcLa_f y rbcLa_rev funcionan adecuadamente en nuestras condiciones de trabajo y para las especies estudiadas.
En cuanto a la secuenciación de los productos de amplificación de rbcL, se obtuvieron secuencias de buena calidad en el 83% de las muestras de referencia. El largo promedio de las secuencias fue de 575 bases, siendo muy similar en todas las especies analizadas. En el caso de las muestras comerciales industrializadas, el 50% de las secuencias obtenidas fueron de calidad aceptable. Las muestras de albahaca y estragón debieron ser amplificadas y secuenciadas en una segunda instancia, con lo que se logró mejorar la calidad.
Los resultados obtenidos para la región rbcL avalan las conclusiones del estudio realizado por el grupo de plantas del CBOL en las especies estudiadas en este trabajo. Este locus es fácilmente amplificable con los cebadores disponibles, lo que confirma su universalidad, y las secuencias generadas son de buena calidad en general, requiriendo poca edición manual (CBOL Plant Working Group, 2009).
Con el protocolo puesto a punto para la amplificación de la región matK se analizaron 29 muestras de referencia pertenecientes a las familias Amaryllidaceae, Apiaceae, Astereaceae, Lamiaceae, Verbenaceae. En el 86% de los casos se obtuvieron productos de amplificación del tamaño esperado. La amplificación falló en las especies Origanum majorana, Origanum vulgare y Ocimum pertenecientes a la familia de las Lamiaceas. En estos casos se repitió la amplificación partiendo de la mitad de la concentración de ADN (100 ng), obteniéndose amplicones del tamaño esperado. Al amplificar el ADN de las muestras comerciales no se obtuvieron productos de amplificación para la muestra de arándano. Con las muestras de albahaca y orégano fue necesaria una segunda amplificación.
Las secuencias matK obtenidas para las muestras de referencia fueron de mediana y baja calidad en el 37% de los casos. En todas las secuencias se observó una pérdida de resolución alrededor de las 500 bases. El largo promedio fue de 793 bases, observándose mucha variación en la longitud de las secuencias entre muestras. A partir de los tres amplicones originados de las tres muestras de Ocimum sp, se obtuvieron secuencias para dos muestras, las cuales debieron ser amplificadas y secuenciadas nuevamente para mejorar la intensidad de señal y calidad de las secuencias. Esta situación se repitió con las secuencias de una muestra de Thymus vulgaris y la de Petroselinum crispum. A partir del fragmento amplificado de una de las muestras de Origanum vulgare no se obtuvo secuencia. Las secuencias obtenidas en el 35% de las muestras comerciales fueron de calidad aceptable. El amplicón obtenido a partir de la muestra de albahaca fue secuenciado nuevamente, pero no se logró mejorar la calidad del resultado.
Varios autores han reportado dificultades en la amplificación y la obtención de secuencias bidireccionales de buena calidad de la región matK en distintas especies (Fazekas, et al., 2008; Kressy Erickson, 2007; Hollingsworth, et al., 2011; Theodoridis, et al., 2012). Este hecho fue corroborado en el estudio, observándose dificultades para la amplificación y secuenciación de esta región, tanto en las muestras de tejido fresco como en las procesadas. A pesar de ello, matK sigue siendo considerada como región de referencia junto a rbcL por el CBOL Plant Group, debido a que es una de las regiones codificantes del genoma plastídico que evoluciona más rápidamente, lo que le da un gran poder de discriminación entre especies (CBOL Plant Working Group, 2009; Hollingsworth, et al., 2011).
Por otra parte, las dificultades encontradas con la amplificación y secuenciación de rbcL y matK, sobre todo en muestras comerciales, podría resolverse utilizando la estrategia del mini-barcoding. Se basa en la amplificación de fragmentos más pequeños (<200 pb), lo cual resolvería los inconvenientes que se presentan en los productos botánicos industrializados en los que la estructura del ADN puede estar alterada debido al procesamiento de los tejidos vegetales (Gao, et al., 2019).
Con las secuencias bidireccionales obtenidas y editadas manualmente, se generó una secuencia consenso para cada muestra y cada locus. Con ellas se llevó a cabo la identificación en la base de datos de Barcode of Life (BOLD) de cada marcador por separado, ya que el motor de identificación de BOLD no está diseñado para utilizar ambos marcadores en la misma instancia, pese a que se recomienda el uso de dos loci para plantas. Se reportó la coincidencia más cercana, el/los vecinos más cercanos de igual género (VMCIG), y el/los vecinos más cercanos pertenecientes a otros géneros (VMCDG), clasificados según su score y porcentaje de identidad con la secuencia problema. El criterio utilizado para considerar que la secuencia correspondiente a la muestra de referencia fuera correctamente asignada a la especie a la que pertenece, fue que la misma obtuviera el mayor score entre las noventa y nueve coincidencias reportadas por la base de datos. Se consideró una falla en la asignación cuando la especie correcta obtuvo un score menor o igual al de las otras especies. En los casos en que la diferencia entre los score no era significativa (menor o igual a 2), se tomó en cuenta el porcentaje de identidad para reforzar el criterio. Por último, se informó la especie/género asignado de acuerdo con los datos obtenidos. En las Tablas 4 y 5 se reportan los resultados obtenidos a partir de las muestras utilizadas como referencia. Se unificaron los resultados de las tres muestras analizadas de cada especie para que fuera más sencilla la interpretación de las tablas.
En las Tablas 6 y 7 se presentan los resultados obtenidos de la comparación en BOLD de las secuencias obtenidas para las muestras comerciales para ambos marcadores. En este caso se analizaron los resultados de la identificación con el mismo criterio que para las muestras de referencia, pero en las comerciales no se corroboró la especie declarada por el fabricante mediante una identificación taxonómica. Por este motivo, con la identificación obtenida mediante la comparación de secuencias en BOLD, corroboramos si la misma coincidía o no con la especie declarada por el fabricante.
De los 29 haplotipos rbcL obtenidos para las muestras de referencia, 12 pudieron asignarse correctamente a nivel de especie, 10 a nivel de género y 7 a nivel de familia. Esto representa un 41,4% de asignación correcta a nivel de especie.De los 27 haplotipos matK obtenidos para las muestras de referencia, 14 pudieron asignarse correctamente a nivel de especie, 4 a nivel de género y 9 a nivel de familia. Esto representa un 51,9 9% de asignación correcta a nivel de especie. En la Figura 1 se pueden observar los porcentajes de acierto en la identificación por región, tanto a nivel de especie, género o aquellos ejemplares que no pudieron identificarse.
Tabla 4. Clasificación de muestras de referencia mediante BOLD usando secuencias del locus rbcL (%ID: porcentaje de identidad, VMCIG: vecino más cercano de igual género, VMCDG: vecino más cercano de distinto género, *: no se encontraron vecinos más cercanos de distinto género, **: no se pudo asignar especie ni género).
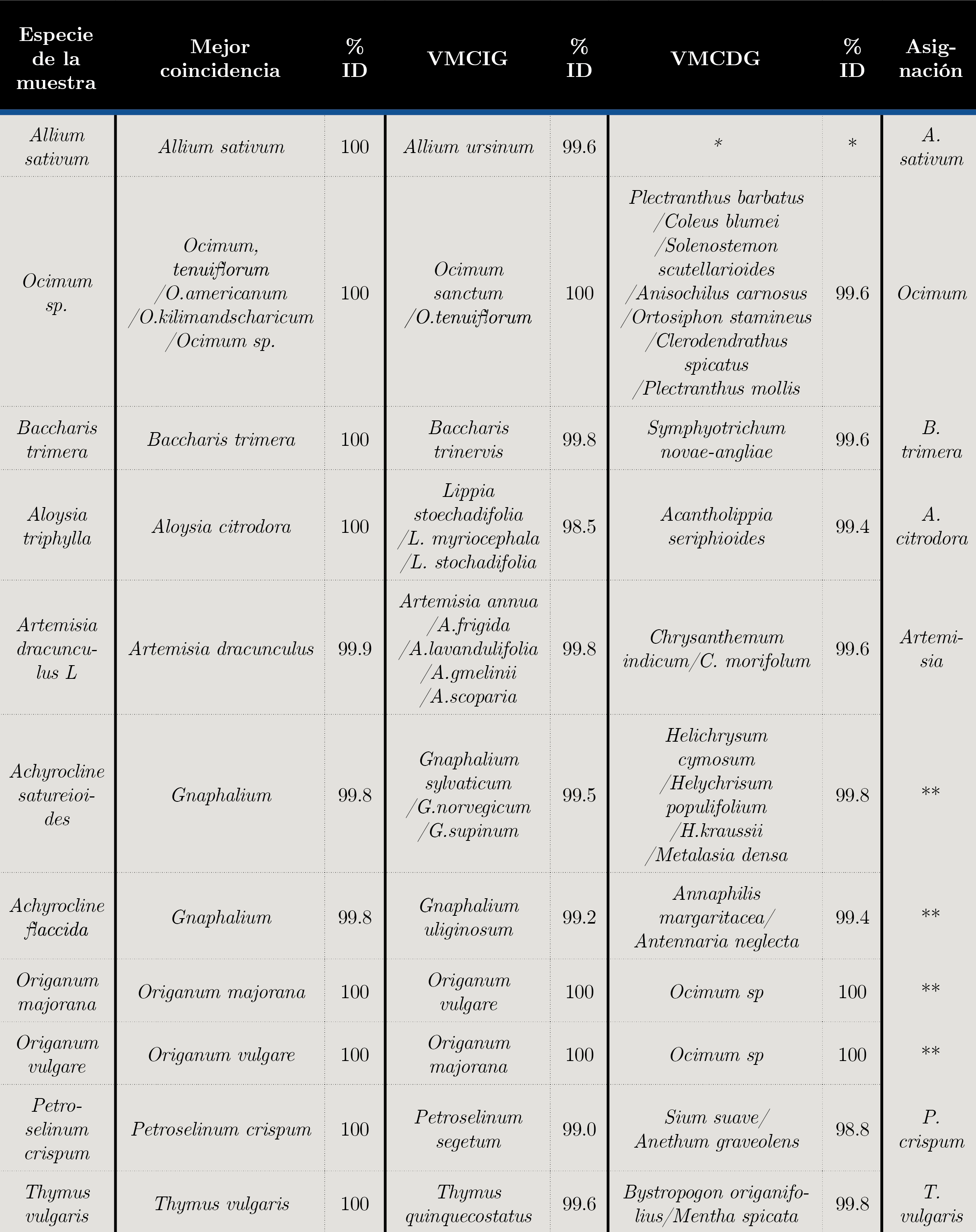
Tabla 5. Clasificación de muestras de referencia mediante BOLD usando secuencias del locus matK (%ID: porcentaje de identidad, VMCIG: vecino más cercano de igual género, VMCDG: vecino más cercano de distinto género, *: no se encontraron vecinos más cercanos de distinto género, **: no se pudo asignar especie ni género).
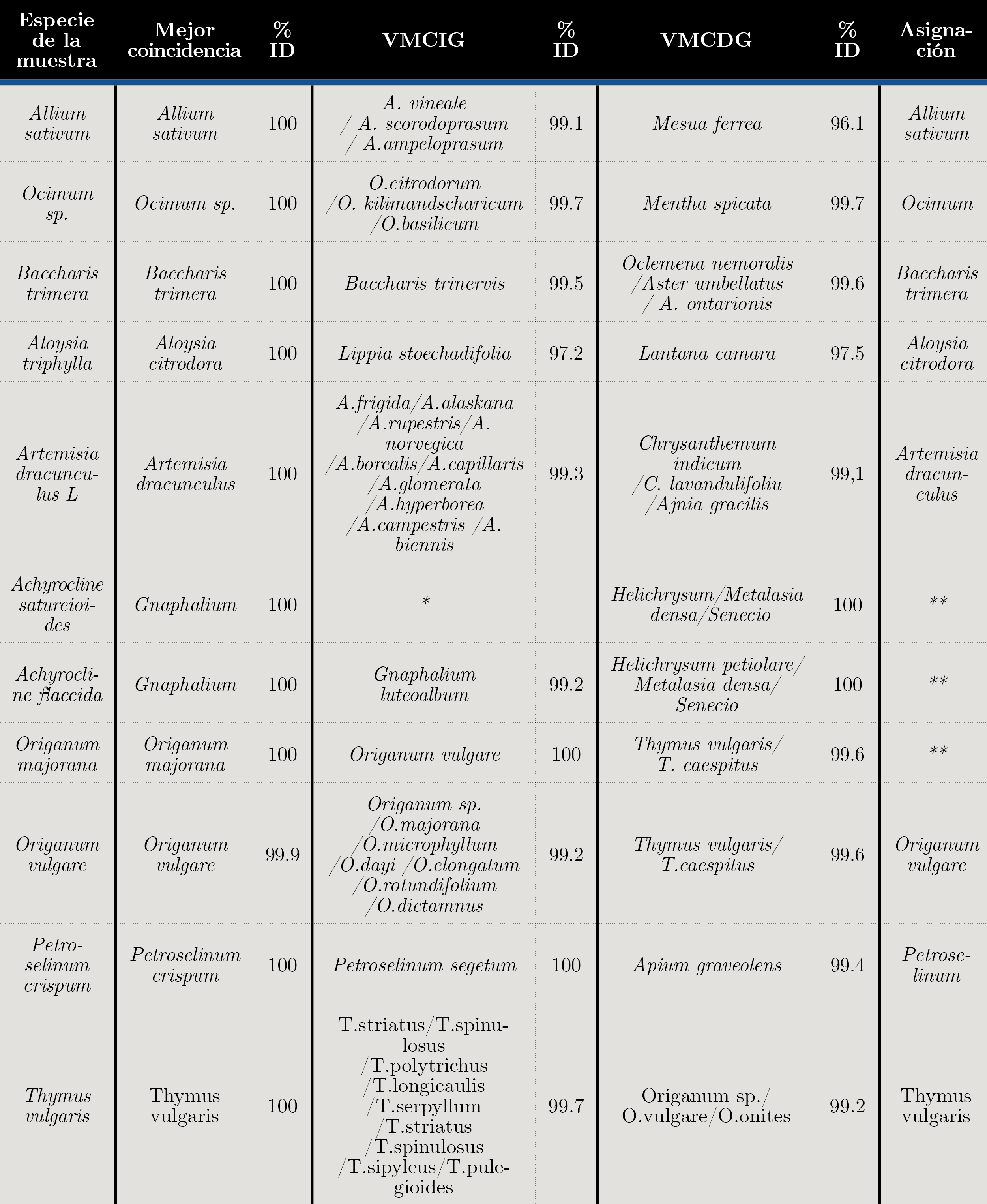
Estos resultados concuerdan con lo expuesto por varios autores con respecto al poder discriminatorio de rbcL con respecto a matK. rbcL es un marcador muy utilizado en estudios filogenéticos de plantas por su robustez para ubicar un espécimen no identificado en la familia, el género y en algunos casos la especie a la que pertenece. Su poder discriminatorio es menor, especialmente en aquellos géneros ricos en especies como ocurre en angiospermas (Kress y Erickson, 2007) y (CBOL Plant Working Group, 2009).
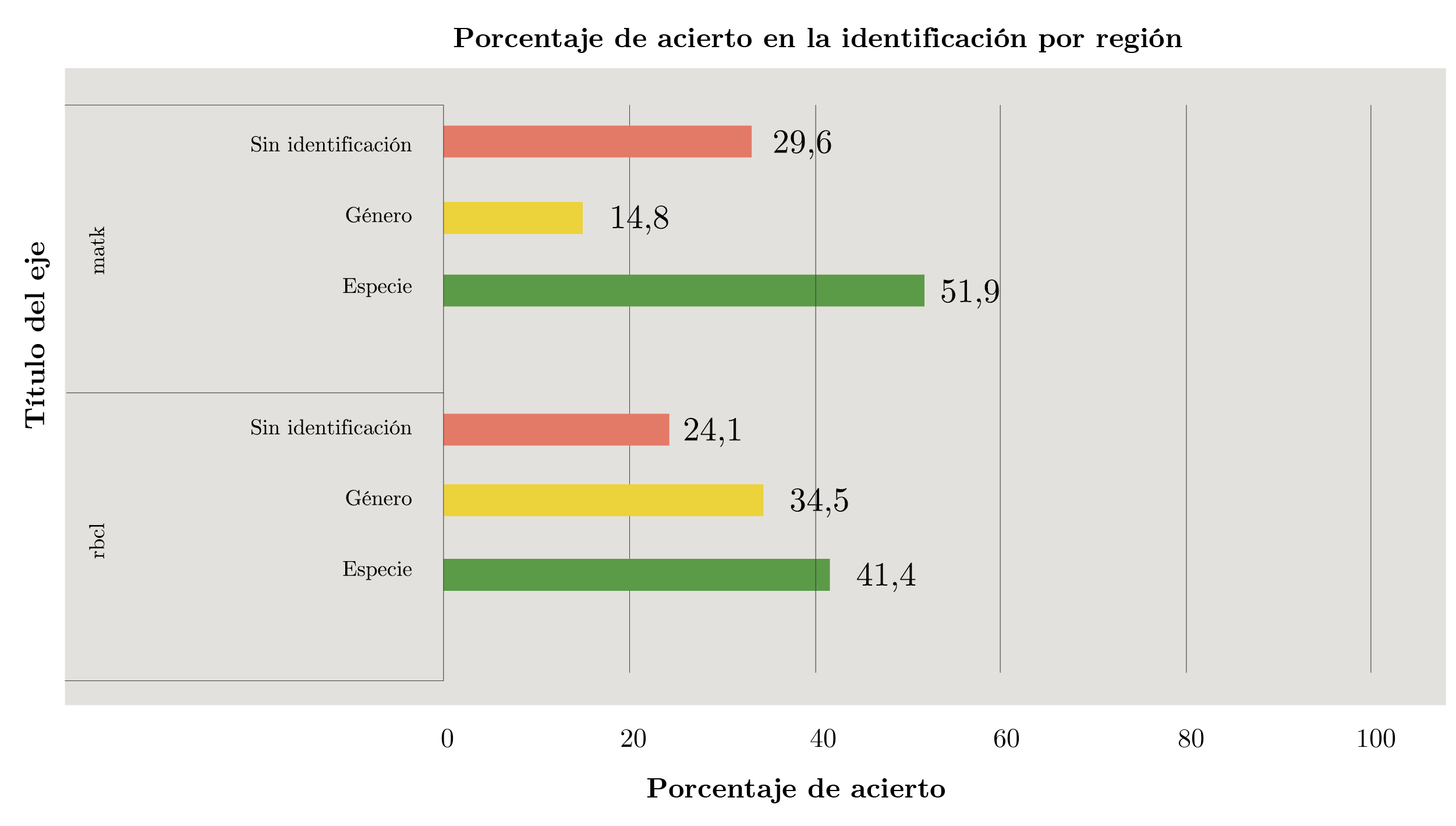
Figura 1. Porcentaje de acierto en la identificación de las muestras de referencia utilizando rbcLo matK
El mismo procedimiento fue realizado con las muestras comerciales. Los resultados obtenidos se presentan en las Tablas 6 y 7. Como puede observarse, con ambos marcadores la mejor coincidencia para la muestra de ajo molido corresponde a la especie declarada por el fabricante. Lo mismo sucedió con el ajo en escamas con el marcador matK. La muestra de estragón no pudo asignarse correctamente usando el marcador rbcL, pero con matK la asignación coincidió con la especie declarada. Con la muestra de perejil la situación fue la inversa, rbcL fue más eficiente para permitir la asignación que matK. El brócoli en cápsulas pudo asignarse correctamente a nivel de género, pero no de especie. Tanto la albahaca como el orégano no pudieron diferenciarse de otras especies pertenecientes a la familia de las Lamiaceas mediante el uso de estos marcadores.
Tabla 6. Clasificación de muestras comerciales mediante BOLD usando secuencias del marcador rbcL (%ID: porcentaje de identidad, VMCIG: vecino más cercano de igual género, VMCDG: vecino más cercano de distinto género).
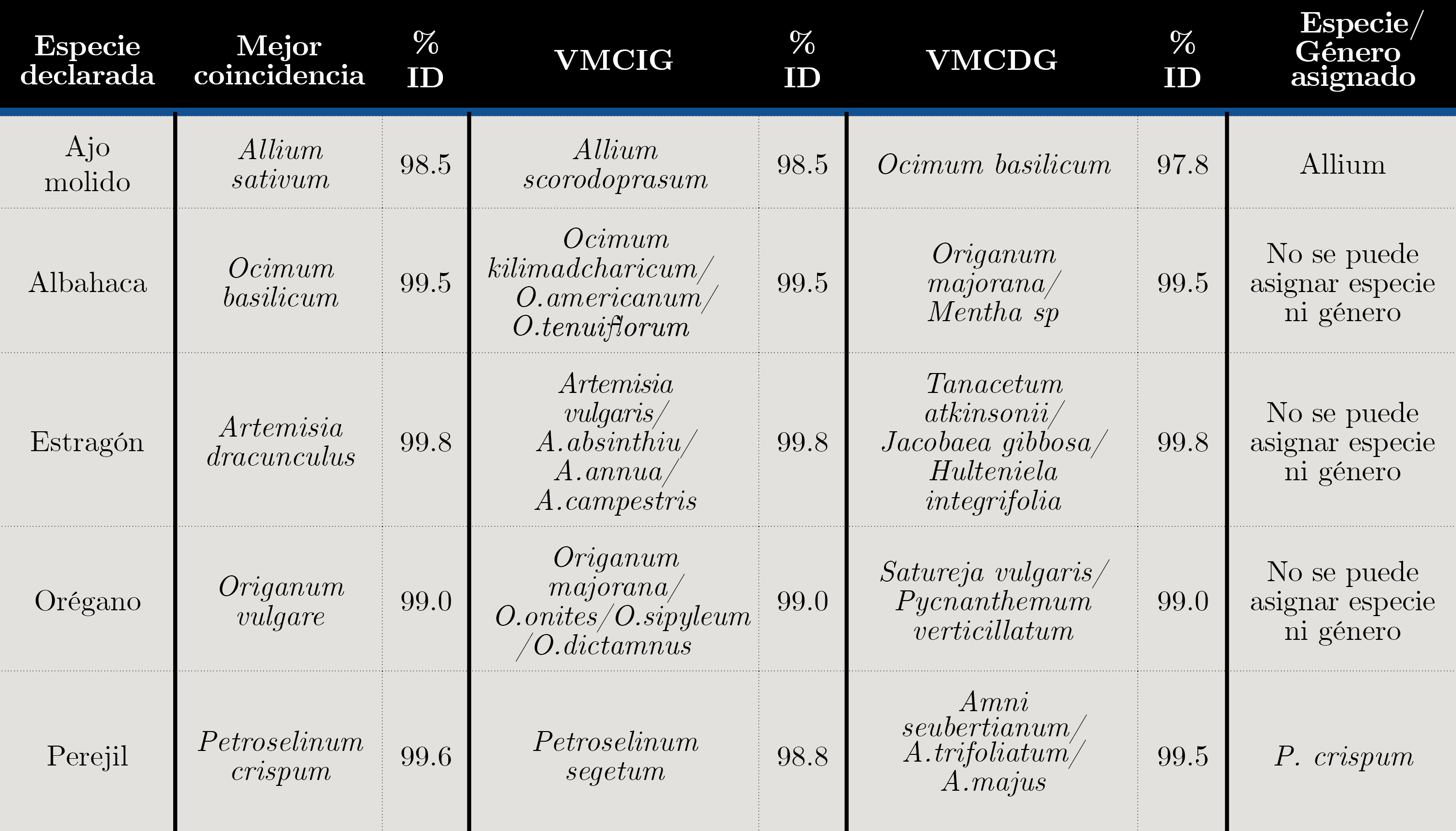
Tabla 7. Clasificación de muestras comerciales mediante BOLD usando secuencias del marcador matK (%ID: porcentaje de identidad, VMCIG: vecino más cercano de igual género, VMCDG: vecino más cercano de distinto género, *: no se encontró mejor coincidencia).

Las secuencias obtenidas para las muestras de referencia y las comerciales fueron utilizadas para construir un dendograma basado en el algoritmo Neighbor Joining y el modelo de cálculo de distancias Kimura 2 Parameter, para cada marcador (Figuras 2 y 3). Esta herramienta se utilizó como forma de validar el agrupamiento de las muestras en conjuntos o clusters a partir de sus secuencias barcode.
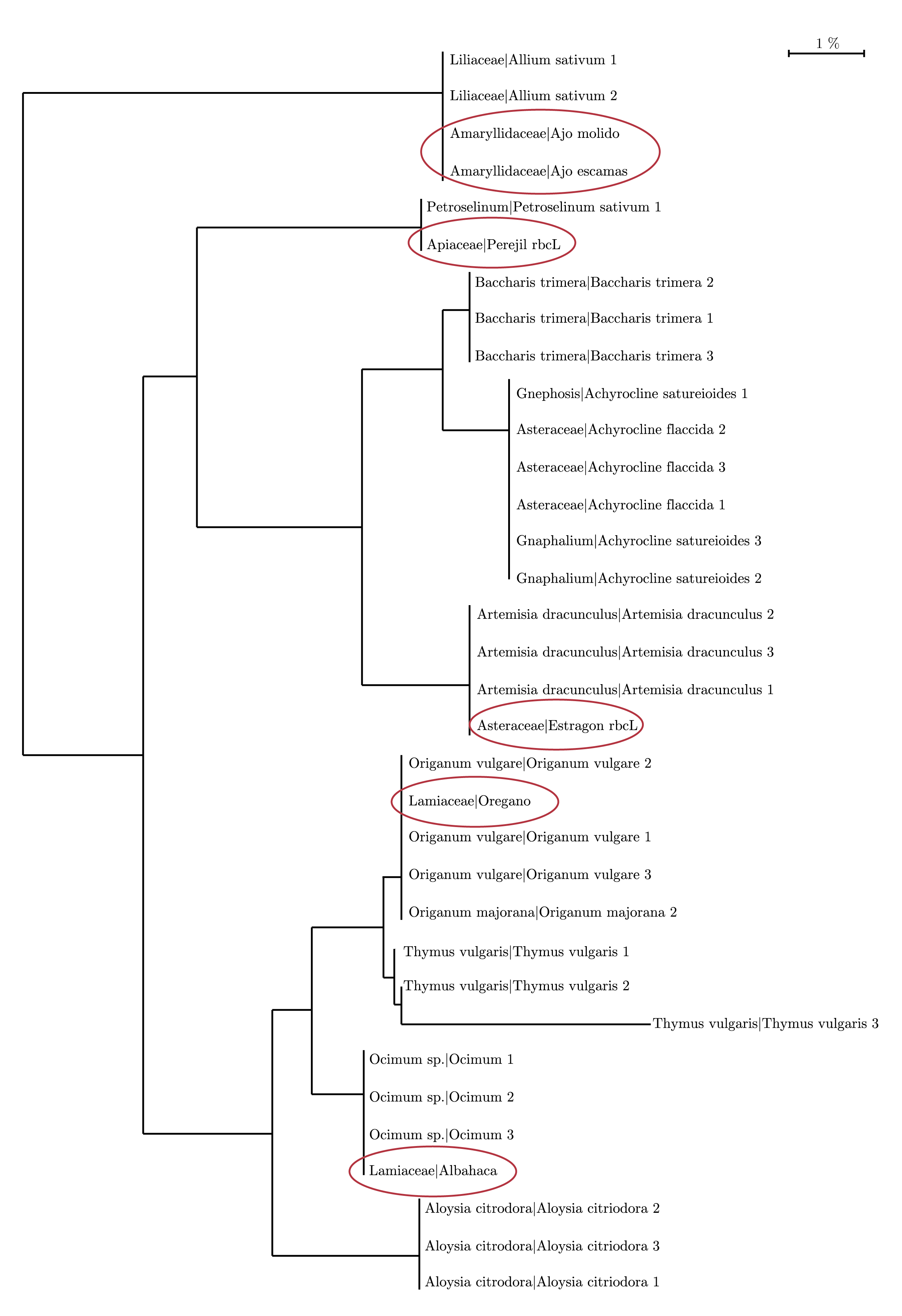
Figura 2. Dendograma en el algoritmo Neighbor Joining y el modelo de cálculo de distancias Kimura 2 Parameter construido a partir de las secuencias rbcL de las muestras de referencia y comerciales. Los círculos rojos señalan la ubicación de las muestras comerciales en el clusters correspondiente.
Las secuencias rbcL se agruparon correctamente por género (Figura 2). En aquellos géneros donde se analizó más de una especie, como Achyrocline y Origanum, las distintas especies fueron agrupadas en el mismo cluster. Las secuencias correspondientes a las muestras comerciales coincidieron con los clusters de las especies declaradas en cada caso.
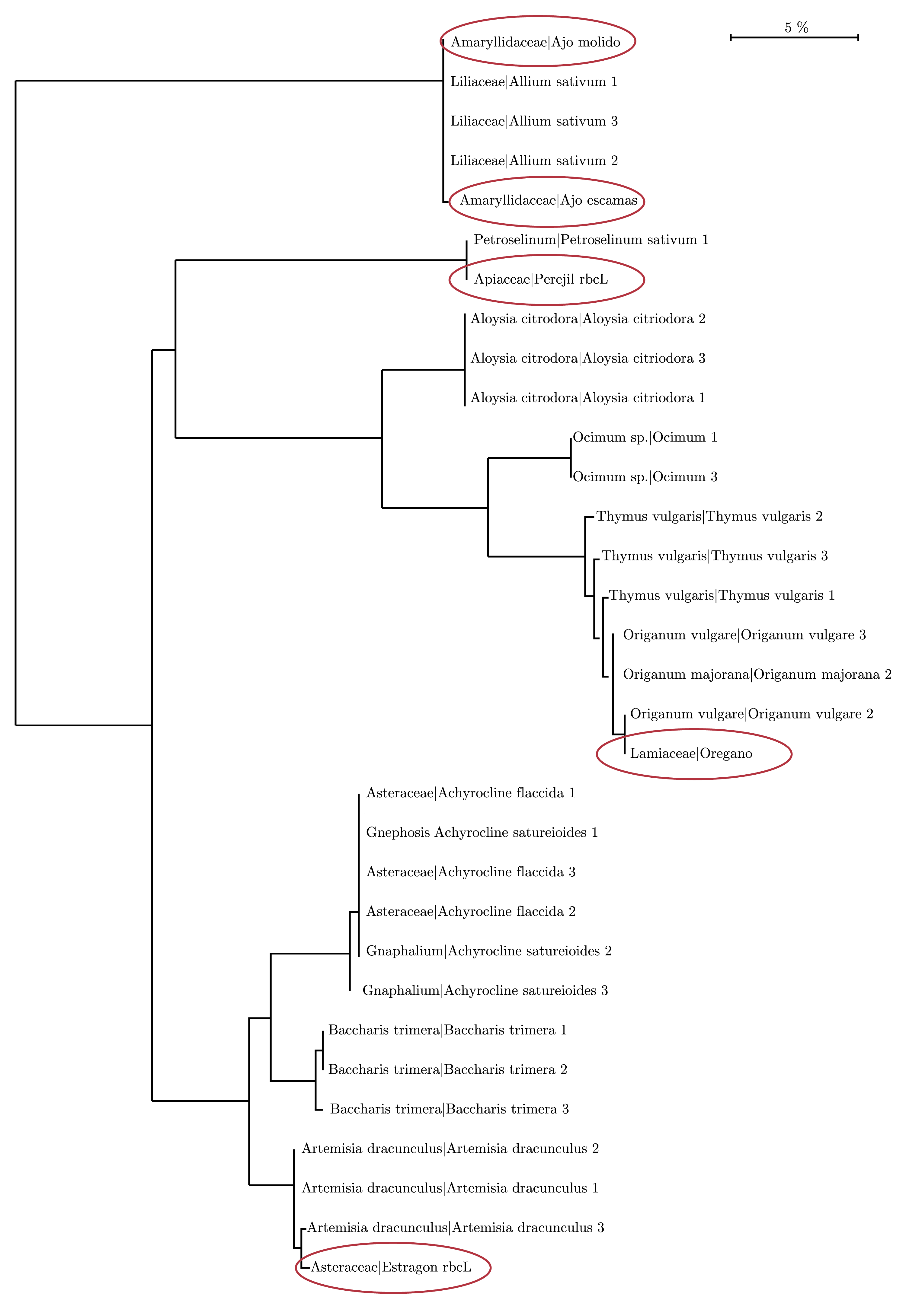
Figura 3. Dendograma basado en el algoritmo Neighbor Joining y el modelo de cálculo de distancias Kimura 2 Parameter construido a partir de las secuencias matK de las muestras de referencia y comerciales. Los círculos rojos señalan la ubicación de las muestras comerciales en el clusters correspondiente.
Las secuencias matK de las muestras de referencia se agruparon de la misma forma que fue descripta para las secuencias rbcL (Figura 3). Las secuencias fueron agrupadas en clusters separados por género, excepto las correspondientes a Origanum y Thymus que comparten el mismo cluster. Las secuencias correspondientes a las muestras comerciales fueron agrupadas en los clusters correspondientes a las especies declaradas.
Estos resultados son concordantes con los presentados en las Tablas 4 y 5, es decir que la asignación a la especie correcta en las muestras analizadas es relativamente baja, y que la discriminación de muestras pertenecientes a especies muy cercanas no fue lograda.
Fazekas y colaboradores (2009) reportaron que la habilidad de las regiones barcode para discriminar entre especies es menor en plantas que en animales, lo cual estaría asociado al hecho de que los límites entre especies son más difusos en las primeras. Esto se debe a fenómenos que ocurren en plantas, como la reproducción asexual, poliploidía e hibridación. En estudios realizados encontraron que las especies monofílicas bien soportadas son menos comunes en plantas, y que la brecha entre distancias genéticas intra e interespecíficas es menos pronunciada que en animales. Como resultado de estos hechos, discriminar entre especies de plantas usando barcode simples o multilocus a partir del genoma plastídico sigue siendo un desafío (Fazekas, et al., 2009).
Según Hollingsworth y otros (2011), para que el Código de Barras de ADN funcione correctamente en plantas se requiere que haya transcurrido suficiente tiempo desde la especiación, a fin de que las mutaciones o la deriva conduzcan a un grupo de caracteres que agrupen a individuos pertenecientes a la misma especie. En clados en los que la especiación es muy reciente o las tasas de mutaciones son muy bajas, es muy probable que las secuencias barcode se compartan entre especies relacionadas.Un ejemplo de esta situación se da en el género Origanum. En nuestro estudio lo incluimos por la importancia de sus especies en el uso culinario, cosmético y medicinal, y con el objetivo de diferenciar entre dos especies muy cercanas: Origanum vulgare y Origanum majorana. Los resultados que obtuvimos concuerdan con las conclusiones de un trabajo realizado por De Mattia y otros (2011). Este es un género que muestra una gran promiscuidad genética. En general, se dan eventos de hibridación entre distintas especies, por lo que la diversidad genética intraespecífica es mayor que la interespecífica, lo que hace que el enfoque del barcoding no sea aplicable ya que no permite discriminar entre ambas especies (De Mattia, et al., 2011). Thymus es otro género en el que las diferencias entre taxones muy relacionados se limitan a pocos caracteres, lo que hace que sean difíciles de asignar a una especie (Galimberti, et al., 2014; Federici, et al., 2015).
Conclusiones
Con el presente trabajo se buscó demostrar la factibilidad de aplicación de técnicas moleculares basadas en ADN conformes con la estrategia del Código de Barras de ADN para la clasificación de muestras de especias y hierbas medicinales, frescas e industrializadas.
De acuerdo con lo que se había planteado, se puso a punto la extracción de ADN de este tipo de muestras, así como la amplificación y secuenciación de regiones barcode propuestas por el grupo de trabajo de plantas del Consortium for the Barcode of Life, rbcL y matK.
Se utilizó la plataforma Barcode of Life Data Systems (BOLD) para validar la clasificación de muestras de referencia de especies conocidas y ubicar muestras problema en grupos de referencia. Se observó que la clasificación de muestras desconocidas es más difícil de lo esperado, especialmente si las muestras de referencia corresponden a poblaciones distintas que las muestras problema.
Una de las principales limitantes es la cobertura a nivel taxonómico de las bases de datos de referencia. Para que esta metodología sea factible de aplicar a nivel industrial, resulta fundamental la existencia de una base de datos global curada que contenga una gran cantidad de secuencias barcode de referencia pertenecientes a distintas poblaciones de las mismas especies. Esto aseguraría una representación de la variabilidad genética dentro de cada especie, haciendo posible la diferenciación de muestras pertenecientes a especies cercanas.
